蛋白质失衡以及细胞与细胞外基质(ECM)相互作用的异常是多种癌症发生和发展的关键因素。癌变的特征是ECM与肿瘤细胞之间的相互作用发生改变。在HPV相关的病变和癌变中,例如宫颈癌,ECM结构和功能的改变很常见。
为了对比健康女性和CIN3患者尿液蛋白质图谱的差异,斯洛伐克帕沃尔•约瑟夫•沙法里克大学Peter Bober团队采用MAILDI-TOF/MS结合LC-MS/MS技术对尿液样本进行无标定量分析,并在《International Journal of Molecular Sciences》(IF=6.208)上发表研究成果《Differential Urinary Proteomic Analysis of High-Risk Cervical Intraepithelial Neoplasia》。研究表明:在CIN3患者中,ECM受体互作通路下调表达,包括13种蛋白(HSPG2, COL6A1, COL6A3, SPP1, THBS1, TNC, DAG1, FN1, COMP, GP6, VTN, SDC1, and CD44)。ECM受体互作通路蛋白质水平降低可能导致细胞与ECM之间的相互作用发生紊乱,并且在宫颈癌的发生和进展中起到重要作用。
 研究者招募10名健康女性和10名CIN3患者,并采集她们的尿样进行检测和分析。采用MALDI-TOF/MS和LC-MS/MS技术检测所有样品的质谱,依靠SwissProt和Mascot数据库用来对质谱数据进行解析,使用Proline软件将图谱进行匹配,从而在组间(健康对照组和CIN3组)进行蛋白鉴定和无标定量分析。此外,研究者利用GSEA软件进行基因集富集分析识别显著富集的通路,其中信号通路的数据来源于KEGG。
研究者招募10名健康女性和10名CIN3患者,并采集她们的尿样进行检测和分析。采用MALDI-TOF/MS和LC-MS/MS技术检测所有样品的质谱,依靠SwissProt和Mascot数据库用来对质谱数据进行解析,使用Proline软件将图谱进行匹配,从而在组间(健康对照组和CIN3组)进行蛋白鉴定和无标定量分析。此外,研究者利用GSEA软件进行基因集富集分析识别显著富集的通路,其中信号通路的数据来源于KEGG。
研究表明:
1. 研究者利用遗传算法(GA)、监督性神经网络(SNN)、快速分类算法(QC)建立分类模型。三种模型的交叉验证值和识别能力如下:
 识别能力最高的GA模型中10个质谱峰重合区域的m/z分别为:2050.8、 2200.6、2291.0、2618.7、3343.6、3377.6、3414.6、6525.5、15,514.5 和18,202.8。在健康对照组和CIN3组中的平均表达量存在差异的质谱范围被放大成如下A-D图:
识别能力最高的GA模型中10个质谱峰重合区域的m/z分别为:2050.8、 2200.6、2291.0、2618.7、3343.6、3377.6、3414.6、6525.5、15,514.5 和18,202.8。在健康对照组和CIN3组中的平均表达量存在差异的质谱范围被放大成如下A-D图:
 2. MALDI-TOF/MS的聚类分析显示:利用质荷比2618.7(m/z)和质荷比3010.0(m/z)的多肽/蛋白质可以很好的对健康女性和CIN3患者进行区分。其中质荷比为2618.7(m/z)的多肽/蛋白质ROC曲线的AUC面积为0.98。
2. MALDI-TOF/MS的聚类分析显示:利用质荷比2618.7(m/z)和质荷比3010.0(m/z)的多肽/蛋白质可以很好的对健康女性和CIN3患者进行区分。其中质荷比为2618.7(m/z)的多肽/蛋白质ROC曲线的AUC面积为0.98。

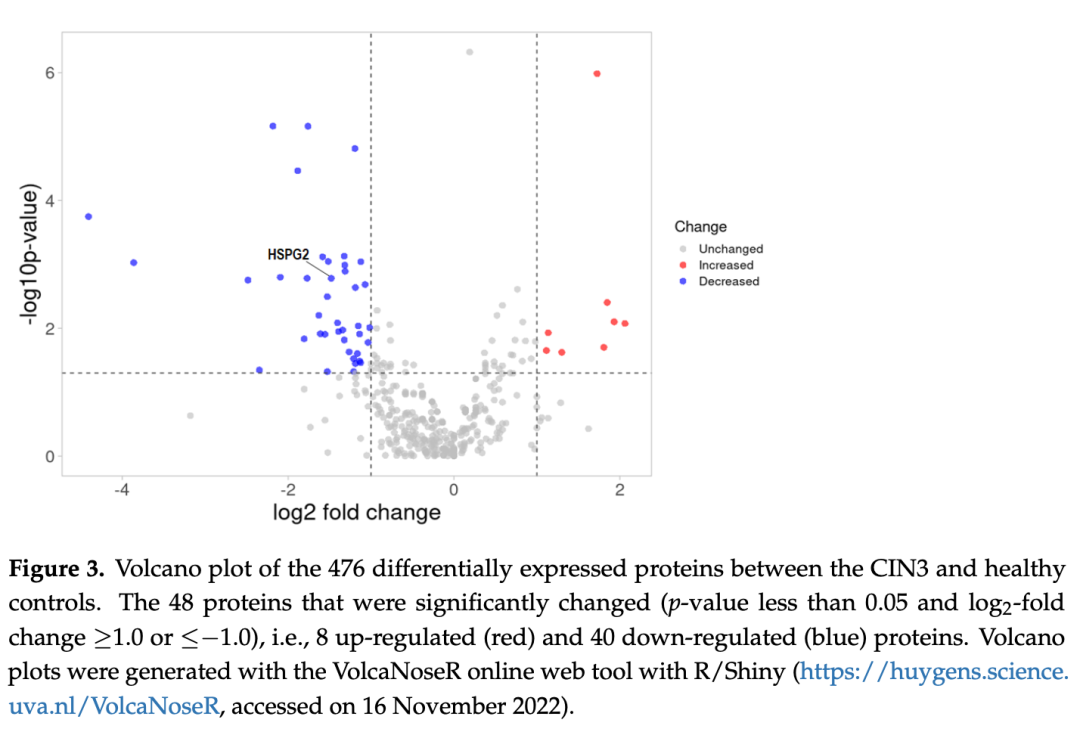
3. 无标定量分析显示:在476种已鉴定的蛋白质中,与健康对照组相比,CIN3组有48种蛋白质发生显著改变,其中40种蛋白的表达水平显著下调,8种显著上调。
4. 研究者通过基因集富集分析发现了在ECM受体互作通路中存在富集的基因集,热图展示了ECM受体互作通路下16个核心基因集在所有样本中表达量的分布。
 在ECM受体互作通路中一共有88种蛋白质,在Pathview Web Server 中15种展现了调节变化。在CIN3组中,13种蛋白质的表达被下调(HSPG2、COL6A1、COL6A3、SPP1、THBS1、TNC、DAG1、FN1、COMP、GP6、VTN、SDC1、CD44; log2 FC[-0.03—-1.48]),2种蛋白质的表达被上调(TNXB、COL4A2; log2 FC[0.09—0.14])。其中,HSPG2的变化是显著的。
在ECM受体互作通路中一共有88种蛋白质,在Pathview Web Server 中15种展现了调节变化。在CIN3组中,13种蛋白质的表达被下调(HSPG2、COL6A1、COL6A3、SPP1、THBS1、TNC、DAG1、FN1、COMP、GP6、VTN、SDC1、CD44; log2 FC[-0.03—-1.48]),2种蛋白质的表达被上调(TNXB、COL4A2; log2 FC[0.09—0.14])。其中,HSPG2的变化是显著的。
 MALDI-TOF/MS 结果证实了健康对照组和 CIN3 组之间蛋白图谱的差异,可作为有效区分健康女性和CIN3患者的工具。从ECM受体互作通路分析可以看出,ECM蛋白即HSPG2、COL6A1、COL6A3、SPP1、THBS1、TNC、DAG1、FN1、COMP、GP6、VTN、SDC1、CD44水平的降低,可能引起细胞与 ECM 相互作用的紊乱,在宫颈癌的发生、发展中起重要作用。然而,还需要进一步的研究来充分理解蛋白质如何影响相关疾病中 ECM 的动态平衡。
MALDI-TOF/MS 结果证实了健康对照组和 CIN3 组之间蛋白图谱的差异,可作为有效区分健康女性和CIN3患者的工具。从ECM受体互作通路分析可以看出,ECM蛋白即HSPG2、COL6A1、COL6A3、SPP1、THBS1、TNC、DAG1、FN1、COMP、GP6、VTN、SDC1、CD44水平的降低,可能引起细胞与 ECM 相互作用的紊乱,在宫颈癌的发生、发展中起重要作用。然而,还需要进一步的研究来充分理解蛋白质如何影响相关疾病中 ECM 的动态平衡。
参考文献:
1. Bober P, Tkáčiková S, Talian I, Urdzík P, Toporcerová S, Sabo J. Differential Urinary Proteomic Analysis of High-Risk Cervical Intraepithelial Neoplasia. Int J Mol Sci. 2023 Jan 28;24(3):2531. doi: 10.3390/ijms24032531. PMID: 36768853; PMCID: PMC9916937.

